近日,我中心李金宇/徐芃课题组在Chemical Science发表题为“DSSA-PPI: Enhancing binding affinity change prediction upon protein mutations using disentangled structure-sequence aware attention”的研究论文。该工作提出了一种融合蛋白质结构与序列信息的新型深度学习模型,能够高通量、高精度地预测突变对蛋白质结合亲和力的影响。

蛋白质-蛋白质相互作用(PPI)是生命活动与功能调控的核心机制。明确氨基酸突变如何影响PPI,不仅有助于揭示疾病发生机理、解读突变致病性,也为蛋白质工程与靶向药物设计提供关键依据。尤其是在抗体设计与蛋白优化领域,面对百万甚至千万级别的潜在突变组合,传统实验筛选与计算方法往往力不从心。为此,研究团队创新性地开发了基于解耦注意力机制的多模态深度学习模型DSSA-PPI,该模型协同整合结构与序列信息,实现了对突变引起的结合亲和力变化的高精度预测。
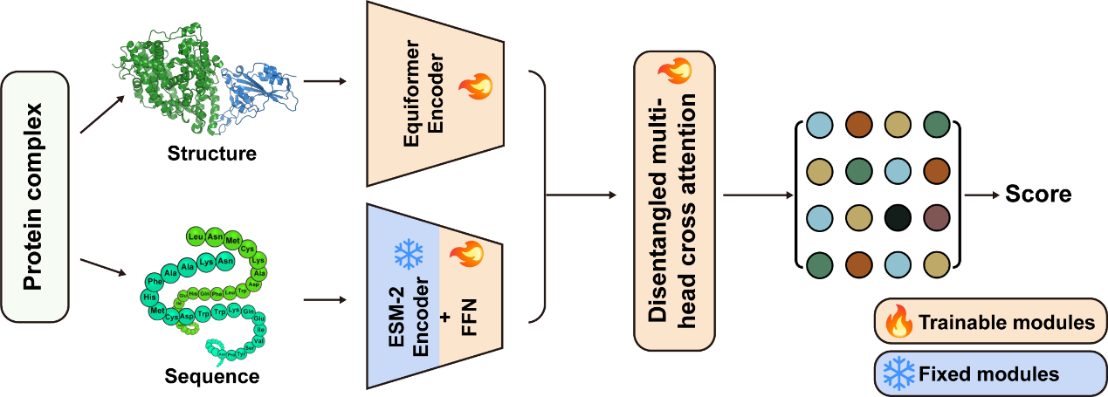
图1 DSSA-PPI工作流程示意图
在多项测试中,DSSA-PPI展现出优秀的鲁棒性与泛化能力,其整体预测性能优于当前主流方法。
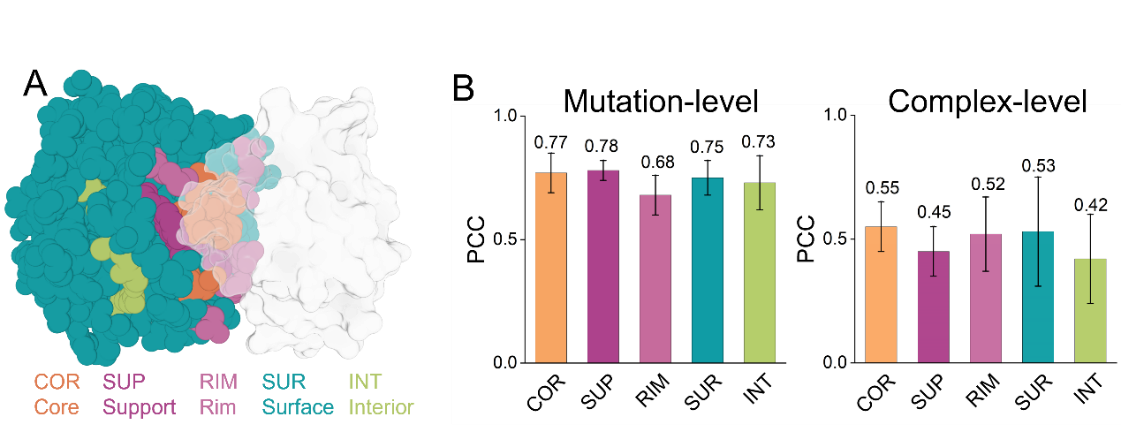
图2 DSSA-PPI模型在不同突变区域下的预测性能
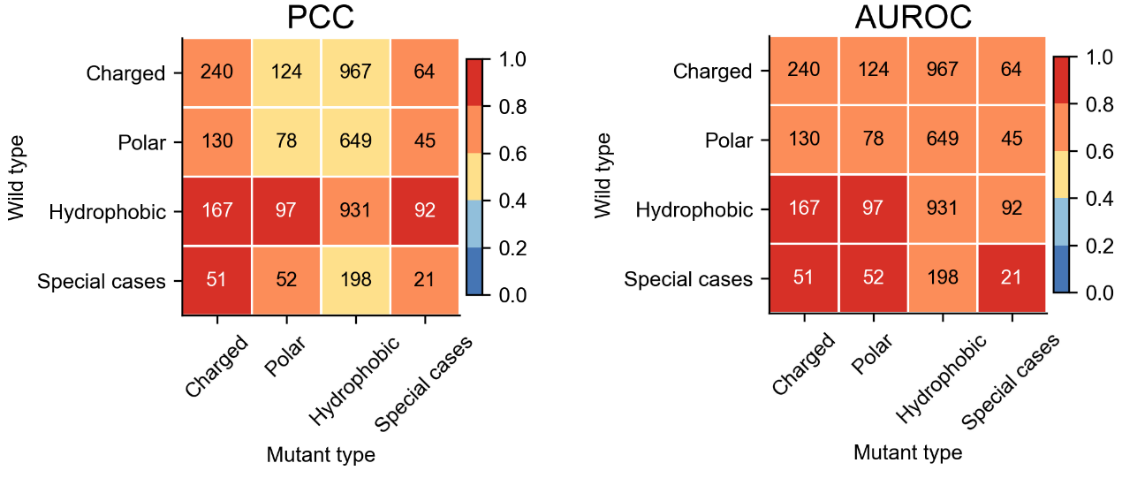
图3 DSSA-PPI模型在不同突变类型下的预测性能
为验证模型的实际应用潜力,团队开展了多个典型案例分析。在SARS-CoV-2刺突蛋白与ACE2相互作用体系中,DSSA-PPI不仅准确识别出增强结合的突变,还成功捕捉到多个“关切变异株”(VOCs)中的关键突变特征,显示出在病毒进化分析与抗疫研究中的应用价值。
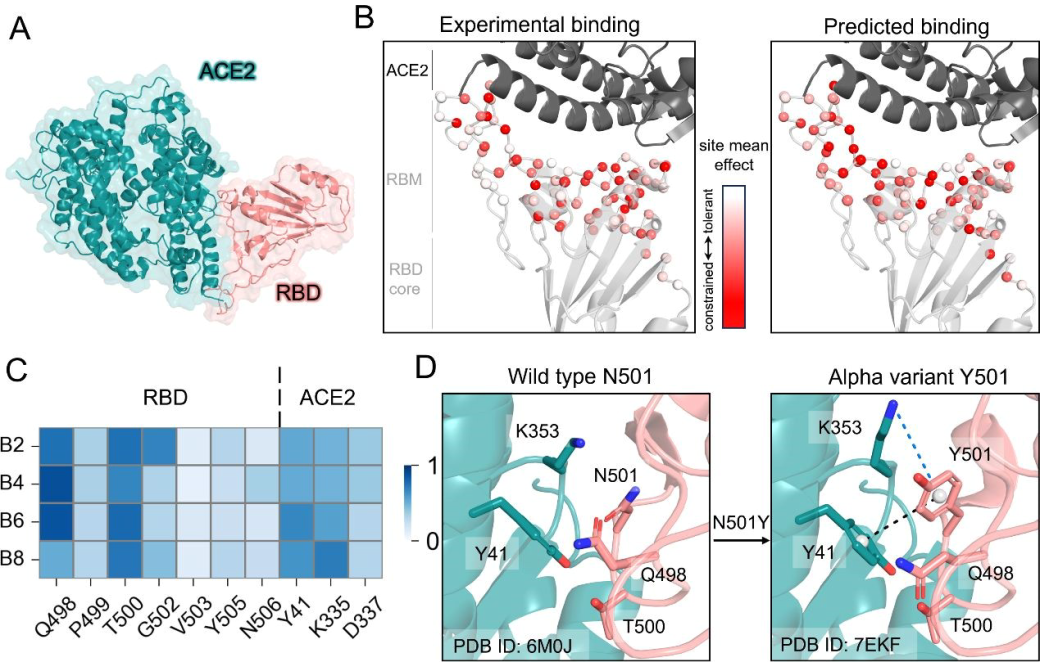
图4 DSSA-PPI在SARS-CoV-2 RBD-ACE2体系中的预测表现与可解释性分析
此外,该模型还被成功应用于活化凝血因子XI(FXIa)环肽抑制剂的亲和力优化,指导了实际的分子改造,进一步证明了其在药物研发与蛋白质工程中的实用性与可靠性。
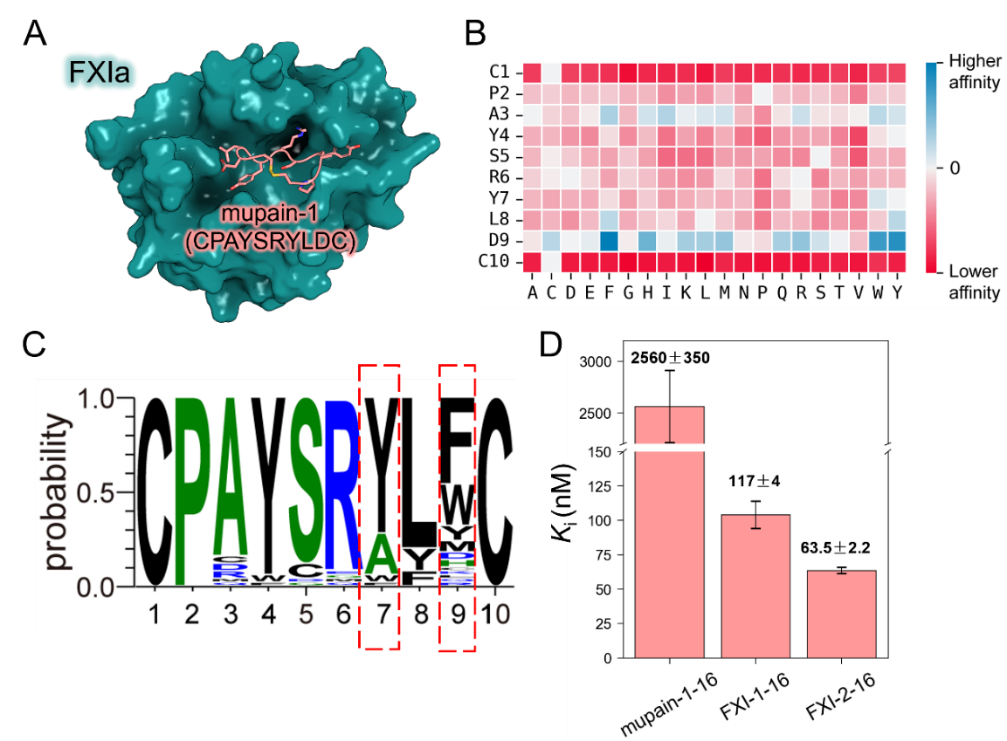
图5 DSSA-PPI辅助环肽抑制剂亲和力优化
综上所述,DSSA-PPI作为一个通用、高效的计算工具,为系统评估突变对蛋白质互作的影响提供了新的解决方案,有望在疾病机制解析、蛋白质工程及创新药物研发等领域发挥重要作用。
论文信息
本研究第一作者为我中心李金宇课题组博士生吴钜洪,李金宇教授与徐芃教授为共同通讯作者。吉林农业科技学院、吉林医药学院提供了重要技术支持。该研究获国家自然科学基金(22573016)资助。
J. Wu, J. Sun, T. Liang, Y. Zhang, H. Zhang, T. Zhang, X. Feng, P. Gao, P. Xu* and J. Li*. DSSA-PPI: Enhancing binding affinity change prediction upon protein mutations using disentangled structure-sequence aware attention. Chem. Sci., 2026, doi: 10.1039/d5sc08898d.
全文链接:https://pubs.rsc.org/en/content/articlelanding/2026/sc/d5sc08898d
代码与程序链接:https://github.com/Agitw/DSSA-PPI